Ausreißer, Hebelwerte und Einfluss
Einführung in Regression mit R

Richie Cotton
Data Evangelist at DataCamp
Rotaugendatensatz
roach <- fish %>%
filter(species == "Roach")
| Art | length_cm | mass_g |
|---|---|---|
| Rotauge | 12,9 | 40 |
| Rotauge | 16,5 | 69 |
| Rotauge | 17,5 | 78 |
| Rotauge | 18,2 | 87 |
| Rotauge | 18,6 | 120 |
| ... | ... | ... |
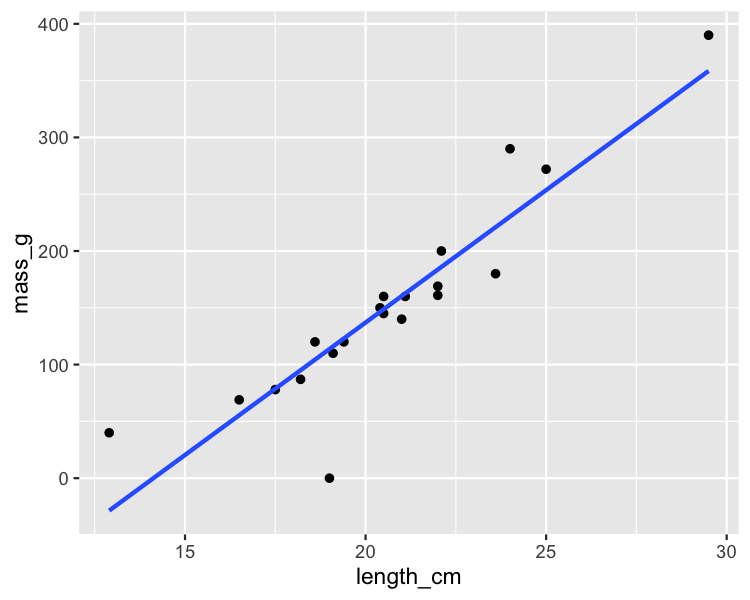
Welche Punkte sind Ausreißer?
ggplot(roach, aes(length_cm, mass_g)) +
geom_point() +
geom_smooth(method = "lm", se = FALSE)
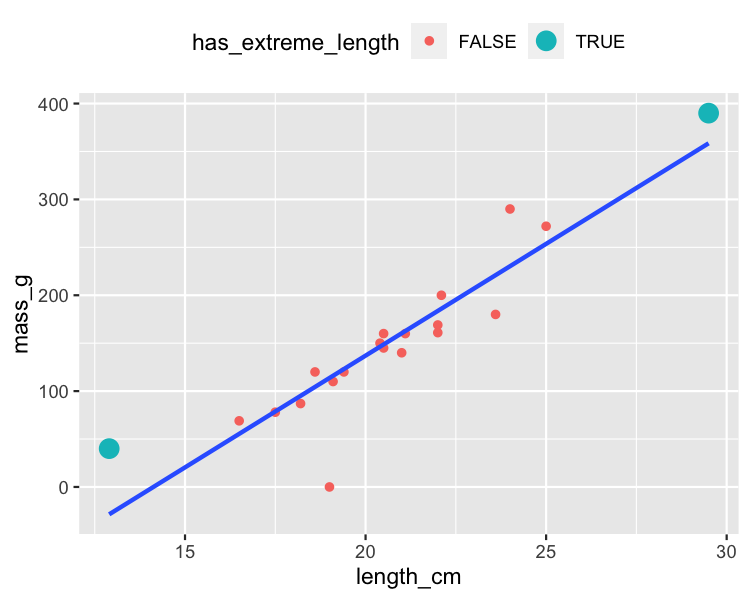
Extreme erklärende Werte
roach %>%
mutate(
has_extreme_length = length_cm < 15 | length_cm > 26
) %>%
ggplot(aes(length_cm, mass_g)) +
geom_point(aes(color = has_extreme_length)) +
geom_smooth(method = "lm", se = FALSE)
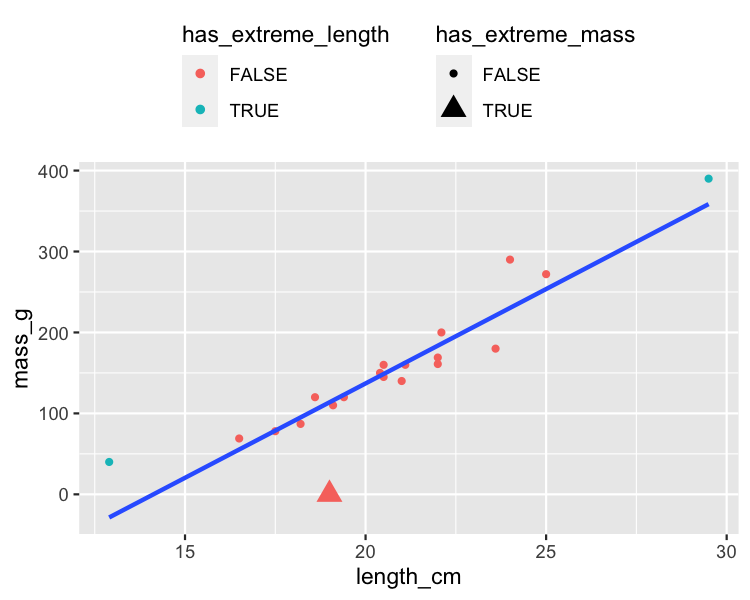
Werte, die von der Regressionsgeraden abweichen
roach %>%
mutate(
has_extreme_length = length_cm < 15 | length_cm > 26,
has_extreme_mass = mass_g < 1
) %>%
ggplot(aes(length_cm, mass_g)) +
geom_point(
aes(
color = has_extreme_length,
shape = has_extreme_mass
)
) +
geom_smooth(method = "lm", se = FALSE)
Hebelwerte
Hebelwert ein Maß dafür, wie extrem die Werte der erklärenden Variablen sind.
mdl_roach <- lm(mass_g ~ length_cm, data = roach)
hatvalues(mdl_roach)
1 2 3 4 5 6 7
0.3137 0.1255 0.0935 0.0763 0.0684 0.0619 0.0605
8 9 10 11 12 13 14
0.0568 0.0503 0.0501 0.0501 0.0506 0.0509 0.0581
15 16 17 18 19 20
0.0581 0.0593 0.0884 0.0995 0.1334 0.3947
Die Spalte „.hat“
library(broom)
augment(mdl_roach)
# A tibble: 20 × 8
mass_g length_cm .fitted .resid .hat .sigma .cooksd .std.resid
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 40 12.9 -28.6 68.6 0.314 33.8 1.07 2.17
2 69 16.5 55.4 13.6 0.126 39.1 0.0104 0.381
3 78 17.5 78.7 -0.711 0.0935 39.3 0.0000197 -0.0196
4 87 18.2 95.0 -8.03 0.0763 39.2 0.00198 -0.219
5 120 18.6 104. 15.6 0.0684 39.1 0.00661 0.424
...
Schaben mit extremen Hebelwerten
mdl_roach %>%
augment() %>%
select(mass_g, length_cm, leverage = .hat) %>%
arrange(desc(leverage)) %>%
head()
# A tibble: 6 x 3
mass_g length_cm leverage
<dbl> <dbl> <dbl>
1 390 29.5 0.395 # really long roach
2 40 12.9 0.314 # really short roach
3 272 25 0.133
4 69 16.5 0.126
5 290 24 0.0995
6 78 17.5 0.0935
Einfluss
Der Einfluss misst, wie stark sich das Modell ändern würde, wenn man eine bestimmte Beobachtung bei der Modellierung aus dem Datensatz weglassen würde.

Cook-Distanz
Die Cook-Distanz ist das gängigste Maß für den Einfluss.
cooks.distance(mdl_roach)
1 2 3 4 5 6
1.07e+00 1.04e-02 1.97e-05 1.98e-03 6.61e-03 3.12e-01
7 8 9 10 11 12
8.53e-04 1.99e-04 2.57e-04 2.56e-04 2.45e-03 7.95e-03
13 14 15 16 17 18
1.37e-04 4.82e-03 1.15e-02 4.52e-03 6.12e-02 1.50e-01
19 20
2.06e-02 3.66e-01
Die Spalte „.cooksd“
library(broom)
augment(mdl_roach)
# A tibble: 20 x 9
mass_g length_cm .fitted .se.fit .resid .hat .sigma .cooksd .std.resid
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 40 12.9 -28.6 21.4 68.6 0.314 33.8 1.07 2.17
2 69 16.5 55.4 13.5 13.6 0.126 39.1 0.0104 0.381
3 78 17.5 78.7 11.7 -0.711 0.0935 39.3 0.0000197 -0.0196
4 87 18.2 95.0 10.5 -8.03 0.0763 39.2 0.00198 -0.219
5 120 18.6 104. 9.98 15.6 0.0684 39.1 0.00661 0.424
...
Die einflussreichsten Rotaugen
mdl_roach %>%
augment() %>%
select(mass_g, length_cm, cooks_dist = .cooksd) %>%
arrange(desc(cooks_dist)) %>%
head()
# A tibble: 6 x 3
mass_g length_cm cooks_dist
<dbl> <dbl> <dbl>
1 40 12.9 1.07 # really short roach
2 390 29.5 0.366 # really long roach
3 0 19 0.312 # zero mass roach
4 290 24 0.150
5 180 23.6 0.0612
6 272 25 0.0206
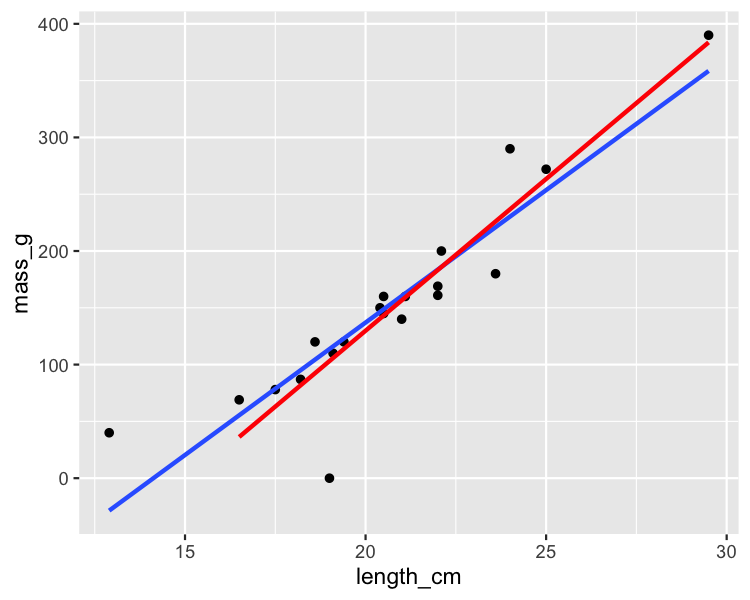
Das einflussreichste Rotauge loswerden
roach_not_short <- roach %>%
filter(length != 12.9)
ggplot(roach, aes(length_cm, mass_g)) +
geom_point() +
geom_smooth(method = "lm", se = FALSE) +
geom_smooth(
method = "lm", se = FALSE,
data = roach_not_short, color = "red"
)
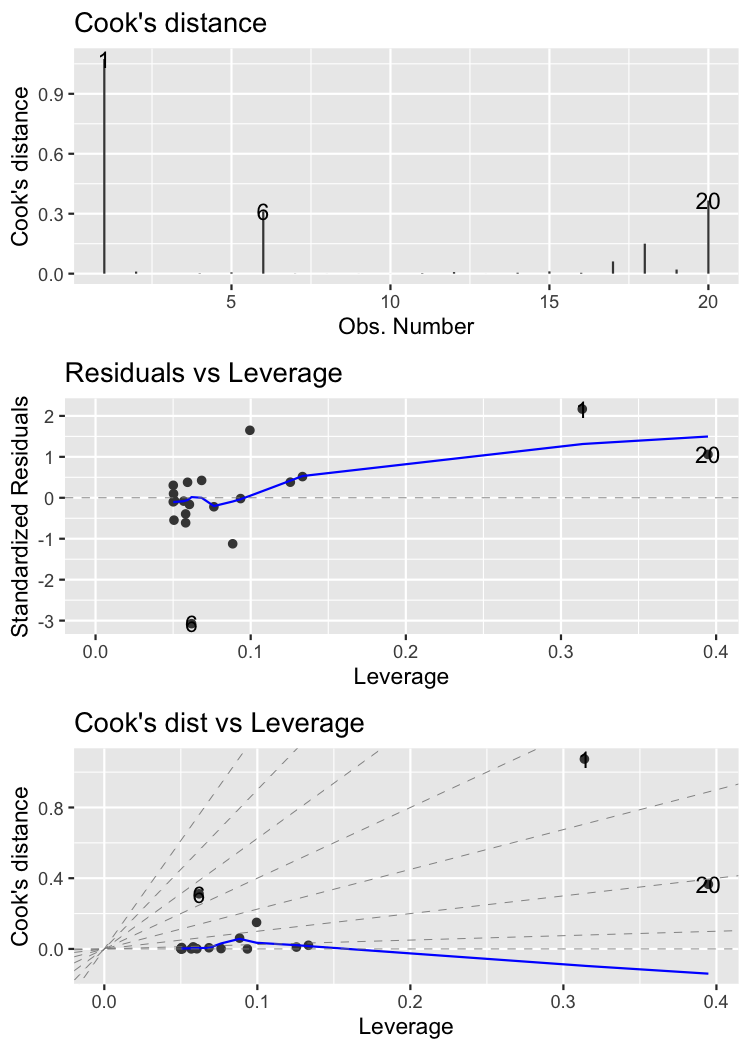
autoplot()
autoplot(
mdl_roach,
which = 4:6,
nrow = 3,
ncol = 1
)
Lass uns üben!
Einführung in Regression mit R

